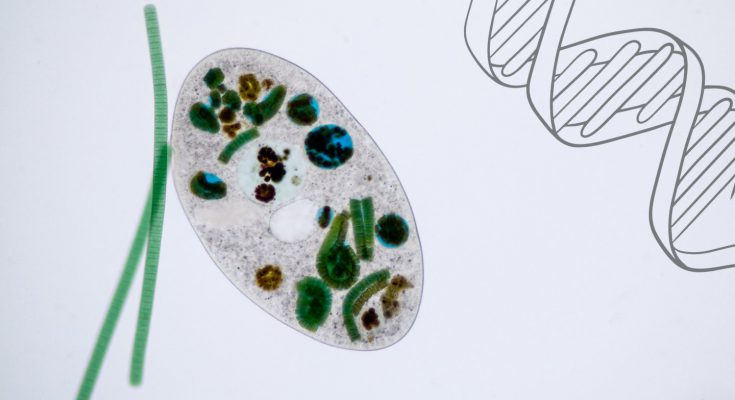
I protisti sono microrganismi eucarioti, generalmente unicellulari, che costituiscono un insieme eterogeneo di circa 50.000 specie diverse. Presentano caratteristiche ecologiche, morfologiche e nutrizionali estremamente diversificate e vengono generalmente classificati in tre sottogruppi: alghe unicellulari, funghi unicellulari e protozoi (Fig. 1).
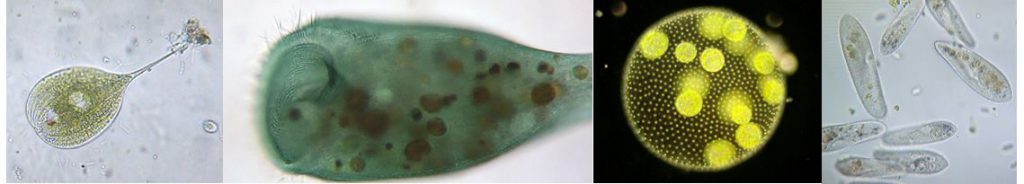
Esempi di protisti.
L’habitat naturale dei protisti è l’acqua allo stato liquido; pertanto, si possono trovare in ambienti di acqua dolce, salata o salmastra, nel sottosuolo o nei terreni umidi, ma anche nei fluidi corporei di altri organismi. Essi si possono ritrovare a vita libera o come simbionti e, a seconda delle caratteristiche metaboliche, si distinguono in organismi autotrofi, eterotrofi o mixotrofi (sia autotrofi che eterotrofi, a seconda delle condizioni ambientali). Generalmente si riproducono per via asessuale mediante diversi meccanismi, quali la scissione binaria o multipla, la gemmazione o la produzione di spore. In particolari condizioni di stress o di modificazioni ambientali, alcuni protisti sfruttano la riproduzione sessuale per dar luogo a nuova variabilità genetica che possa aumentare la probabilità di sopravvivenza delle specie.
Grazie alla loro grande capacità di adattamento hanno colonizzato qualsiasi ambiente terrestre, dalle fredde acque polari alle zone della fascia equatoriale.
La biodiversità caratteristica dei protisti risulta ampiamente sottostimata (Foissner, 2006) per molteplici ragioni, tra cui il poco diffuso interesse verso tali microrganismi e la risoluzione tassonomica delle specie, resa difficile in mancanza di caratteri morfologici univoci. Per questo motivo, è spesso necessario affiancare, alle tradizionali analisi morfologiche, anche quelle di tipo molecolare (Hausmann et al., 2003).
I protisti ciliati, in particolare, sono organismi eterotrofi dotati di estroflessioni membranose, le cilia (Fig. 2). Tali organuli vengono adoperati per la locomozione e la nutrizione e la loro disposizione è generalmente caratteristica per ogni specie. Ulteriore caratteristica distintiva dei ciliati è il dimorfismo nucleare, ovvero la presenza di due diversi tipi di nucleo: un macronucleo e un micronucleo. Il primo, ha funzioni vegetative e dimensioni maggiori rispetto al micronucleo, il quale ha invece funzioni riproduttive.
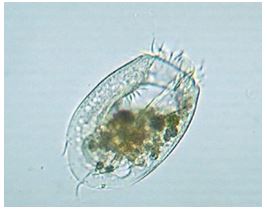
Esempio di protista ciliato (Euplotes).
Risultano descritte circa 4.500 specie di protisti ciliati a vita libera, ma si stima un numero di circa 30.000 specie non ancora descritte (Foissner & Hawksworth, 2009). Le specie di protisti ciliati sono largamente diffuse sul nostro pianeta grazie alla loro notevole capacità di adattarsi a ecosistemi differenti, inclusi gli ambienti estremi e gli ambienti modificati da attività antropiche.
Alcuni protisti ciliati presentano un importante ruolo ecologico come bioindicatori, come marker di cambiamenti climatici passati e, una parte di essi, come patogeni (Pawlowski et al., 2012).
Le grotte sono ambienti sotterranei che sono rimasti relativamente stabili per migliaia di anni e sono spesso caratterizzate dall’assenza di luce, da valori di temperatura dell’aria e dell’acqua costanti, da alti livelli di umidità e limitata disponibilità di nutrienti (Bharti et al., 2022). La composizione dell’aria all’interno delle grotte potrebbe risultare modificata rispetto a quella atmosferica, con alti livelli di gas tossici — quali acido solfidrico (H2S), anidride carbonica (CO2) e metano (CH4) — e bassi livelli di ossigeno (O2).
All’interno delle grotte sono stati trovati organismi adattati a queste condizioni estreme e particolari, caratteristica che probabilmente rende tali organismi più vulnerabili ai cambiamenti climatici. Le gotte rappresentano così un ambiente estremo per la sopravvivenza di specie di microrganismi unicellulari. Pertanto, queste specie potrebbero risultare nuove rispetto alle conoscenze scientifiche del settore, oltre che produrre composti che potrebbero costituire un punto di partenza per la scoperta di nuovi prodotti di origine naturale utilizzabili in vari settori biotecnologici, quali quello farmaceutico o cosmetico.
Lo scopo di questo studio consiste nell’identificazione e caratterizzazione morfologiche e genetiche di protisti ciliati di acqua dolce provenienti da aree ipogee della regione Toscana.
A nostra conoscenza, è il primo studio finalizzato all’identificazione di specie di protisti in grotte della Toscana.
Materiali e metodi
Siti di campionamento e campioni
Sono stati selezionati cinque siti di campionamenti (Figg. 3, 4), distribuiti in diverse aree della Toscana e con caratteristiche geomorfologiche diverse.
Il primo sito di campionamento è rappresentato dalla “Grotta dei Laghi”, situata sui Monti Pisani, nel comune di Agnano (Pisa). La raccolta dei campioni è avvenuta il 4 marzo 2023 con il supporto dell’Associazione Professional Scuba Association International (PSAI) Italia, nelle persone di Leonardo Canale e Alessandro Noferi (Fig. 4): in questo sito sono stati prelevati, in punti diversi, 3 campioni denominati con la sigla “Agn”, dalle lettere inziali del nome del comune in cui è situata la grotta (Agnano).
Il secondo sito di campionamento è rappresentato dalla “Grotta dei Ladri”, situata poco distante dal primo sito e con esso comunicante attraverso piccoli condotti. I campionamenti sono stati effettuati dalla Dott.ssa Luisa Dainelli del Dipartimento di Biologia dell’Università di Pisa il 22 maggio 2023. In questo sito sono stati prelevati, in punti diversi, due campioni, denominati con la sigla “Lad”, dalle lettere del nome della grotta (“dei Ladri”).
Il terzo sito di campionamento è rappresentato dalla “Buca della cima del Monte Matanna”, situata sul Monte Matanna, nel comune di Stazzema (Lucca). In questo sito è stato raccolto un campione il 5 maggio 2023, denominato con la sigla “Mat”, dalle lettere iniziali del Monte (Matanna).
Il quarto sito di campionamento è rappresentato dalla “Grotta n.1 di Poggio Pinzo”, situata sul Monte Argentario, nel comune di Monte Argentario (Grosseto). In questo sito è stato prelevato un campione il 23 aprile 2023, denominato con la sigla “Pin”, dalle lettere iniziali della grotta (Pinzo).
Il quinto ed ultimo sito di campionamento è rappresentato dalla così detta Buca del Pierpi, nome ancora provvisorio in quanto non ancora accastata sul Catasto Grotte Federazione Speleologica Toscana, sita nel comune di Murlo a Siena. All’interno della grotta non c’era acqua, ma una specie di fanghiglia bianca densa. Con opportuni strumenti è stato misurato, all’interno della grotta, un valore del 71% di acido solfidrico, concentrazione estremamente pericolosa per la salute dell’uomo, un valore del 20% di anidride carbonica e un valore dello 0.6% di ossigeno. In questo sito è stato raccolto un campione il 18 giugno 2023, denominato con la sigla “Pie”, dalle lettere iniziali della grotta (Pierpi).
Gli ultimi tre campioni sono stati forniti dal Gruppo Speleologico Torrentistico Speolo (GSTS) in collaborazione con il Gruppo Speleologico Archeologico Apuano e prelevati dagli speleologi Daniele Pagli, Duccio Pieri, Pierpaolo Fiorito, Alessia Fiordaliso, Carlo Ciacci e Giovanni Caponi.
I campioni sono stati prelevati adoperando barattoli di plastica sterili del volume di 100 mL e dotati di tappo.
La metodologia adoperata è quella del “campionamento in sedimenti”: si raccoglie il campione sul fondale del sito, effettuando uno “striscio”, in modo tale che il barattolo risulti riempito per ¼ di volume da substrato, ½ di volume da acqua e ¼ di volume da aria.
Per ciascun sito di campionamento sono stati misurati e annotati parametri fondamentali, quali la temperatura dell’acqua (misurata in situ) e la salinità (misurata in laboratorio) (Tab. 1).
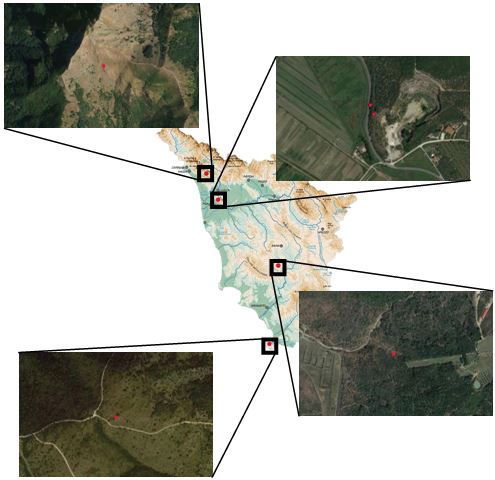
Siti di campionamento; in alto a sinistra il sito Buca della cima del Monte Matanna; in alto a destra i siti Grotta dei Laghi e Grotta dei Ladri; in basso a sinistra il sito Grotta n.1 di Poggio Pinzo; in basso a destra il sito Buca del Pierpi.

Campionamenti della Grotta dei Laghi eseguiti dai subacquei (Leonardo Canale e Alessandro Noferi) della PSAI Italia.
Isolamento e mantenimento in coltura di protisti
Una volta giunti in laboratorio, i campioni sono stati lasciati decantare in modo tale che il substrato si sedimentasse sul fondo del barattolo. Con una pipetta Pauster è stata prelevata un’aliquota di acqua da porre in piastra Petri per eseguire una prima osservazione del campione allo stereo-microscopio; una volta individuate le cellule di protisti, si è proceduti all’isolamento delle stesse.
Con l’utilizzo di una micropipetta sono state isolate le singole cellule osservate, che sono poi state trasferite in vetrini a tre depressioni (Fig. 5a), contenenti una goccia di terreno di coltura, in modo tale che in ciascuna depressione vi fosse una sola cellula. In questa prima fase è stato adoperato un volume ridotto di terreno di coltura (circa 500 μL), in maniera tale da avere, all’osservazione allo stereo-microscopio, un migliore controllo visivo della singola cellula isolata.
I vetrini a tre depressioni così preparati sono stati conservati in una camera umida (Fig. 5b), in maniera tale da evitare l’evaporazione dei liquidi in essi contenuti.

a: Vetrino a tre depressioni. b: camera umida.
Il terreno di coltura utilizzato era costituito da acqua minerale naturale; si tratta di un terreno estremamente versatile, che non richiede preparazione o sterilizzazione e viene comunemente commercializzato in bottiglie.
A distanza di qualche ora dall’isolamento si procedeva con l’alimentazione dei microrganismi, che per i protisti eterotrofi di acqua dolce consiste in inoculi diluiti di una specie di alga verde appartenente al genere Chlorogonium. Si tratta di una specie di protista autotrofo che viene comunemente adoperata per cibare gli eterotrofi di acqua dolce. Gli inoculi di cibo sono stati ripetuti nei giorni successivi per indurre la divisione cellulare ed ottenere così un ceppo (insieme di cellule geneticamente identiche derivanti tutte da divisioni cellulari di una stessa cellula iniziale); la frequenza con cui è stato fornito il cibo e la diluizione di ciascun inoculo dipendevano dalla “salute” (buona motilità e consumo regolare di cibo) delle cellule stesse.
Al raggiungimento di un adeguato numero di cellule per ciascun ceppo isolato, le colture sono state trasferite in provetta, contenitore adoperato per il mantenimento nel tempo delle colture stesse. Ciascuna provetta, corrispondente ad un singolo ceppo, è stata contrassegnata da una sigla formata da 3 lettere che indicavano il sito di campionamento (Agn, Lad, Mat, Pin, Pie), precedute da un numero, corrispondente al campione prelevato, e seguite da un altro numero progressivo, corrispondente al ceppo isolato.
Nella fase di mantenimento in coltura, le provette sono state conservate in celle termostatate a 19 +/- 1 °C.
Analisi morfologica
Le caratteristiche morfologiche dei protisti sono state studiate su cellule vive in modo da osservare la loro forma e struttura. Lo studio è stato condotto mediante l’utilizzo di un microscopio biologico, strumento che consente un’osservazione in vivo delle cellule e, mediante software di acquisizione di immagine, è possibile ottenere immagini e video.
Per osservare adeguatamente i protisti è consigliato rallentare il loro movimento e, a tal scopo, esistono diversi metodi, di tipo meccanico o chimico. In questo studio è stato adoperato un metodo meccanico in cui è stata applicata una delicata pressione sul vetrino copri-oggetto in modo da rallentare il movimento delle cellule fino ad immobilizzarle tra due vetrini (copri-oggetto e porta-oggetto).
Il microscopio utilizzato per le analisi morfologiche è un Optika modello B-383PL con telecamera Optika modello C-B5, equipaggiato con un sistema di acquisizione di immagini digitali.
I successivi step di analisi (identificazione genetica) sono stati condotti solo sui campioni provenienti dal sito della Grotta dei laghi, in particolar modo sono state selezionati 4 ceppi, le cui cellule erano presenti in maggior quantità e si muovevano in modo attivo. I 4 ceppi selezionati per le successive analisi genetiche sono stati conservati senza inoculo di cibo per una settimana, allo scopo di ridurre il più possibile contaminazioni di DNA provenienti dalle microalghe utilizzate come cibo.
Analisi molecolari e identificazione genetica
Per l’estrazione del DNA è stato adoperato il kit commerciale “QIAmp DNA Micro Kit” (Qiagen, Milano, Italia) che è particolarmente indicato per l’estrazione di DNA genomico nel caso di campioni con quantità esigue di materiale, come nella biologia forense per l’analisi di piccoli volumi di sangue o tracce di fluidi corporei.
Come marcatore genetico specie-specifico è stato selezionato uno dei marker genetici più utilizzati in Protistologia per studi di filogenesi molecolare e di caratterizzazione genetica delle specie, cioè il gene nucleare dell’RNA ribosomiale (rRNA) 18S. Il marcatore genetico selezionato è stato amplificato mediante la tecnica della PCR (Polymerase Chain Reaction), utilizzando il primer forward 18S F9 (5′-CTGGTTGATCCTGCCAG-3′) (Medlin et al., 1988) e il primer reverse 18S R1513 Hypo (5′-TGATCCTTCYGCAGGTTC-3′) (Petroni et al., 2002). Le reazioni di amplificazione sono state eseguite aggiungendo aliquote di DNA (100 ng) a una miscela di reazione da 50 μL contenente 2 mM MgCl2, 250 mM dNTP, un’unità di Taq DNA polimerasi (Merk Life Science, Milano, Italia) e 0,2 mM di ciascun primer. Le reazioni di PCR sono state eseguite nello strumento GenAmp System PCR 2400 (Applied Biosystems, Monza, Italia), impostato con i seguenti parametri: una fase di denaturazione iniziale a 94 °C per 1 minuto, seguita da 35 cicli a 94 °C per 30 secondi, a 50 °C per 30 secondi e a 72 °C per 1 minuto, con una fase di estensione finale a 72 °C per 10 minuti. I prodotti di PCR sono stati visualizzati in un gel di agarosio all’1% e le reazioni positive sono state sequenziate con tre primer interni alla sequenza genica amplificata (Tab. 2).
Tab. 2: Denominazione e sequenza dei primer interni usati per sequenziare il gene 18S.

Gli elettroferogrammi ottenuti e corrispondenti alle sequenze geniche (Fig. 6) sono stati verificati con il programma BioEdit (versione 7.0.0) (Hall, 1999) per rilevare eventuali errori di lettura che poi sono stati opportunamente corretti. Le sequenze geniche ottenute sono state confrontate mediante il software BLAST con le sequenze disponibili nel database molecolare GenBank, considerando solo quelle che presentavano le maggiori percentuali di identità di sequenza. Inoltre, le sequenze nucleotidiche ottenute sono state allineate tra loro e con le rispettive sequenze omologhe, derivanti dall’analisi comparativa con il database molecolare, utilizzando il programma Clustal X versione 1.81 (Thompson et al., 1997). Per ciascuna sequenza nucleotidica ottenuta è stata determinata la percentuale in contenuto di GC, ovvero il quantitativo di coppie di basi azotate di guanina e citosina. Tale parametro è correlato alle proprietà fisiche delle molecole di DNA e permette l’identificazione dei corredi genomici delle diverse specie in quanto ognuna ha un suo contenuto in GC caratteristico.
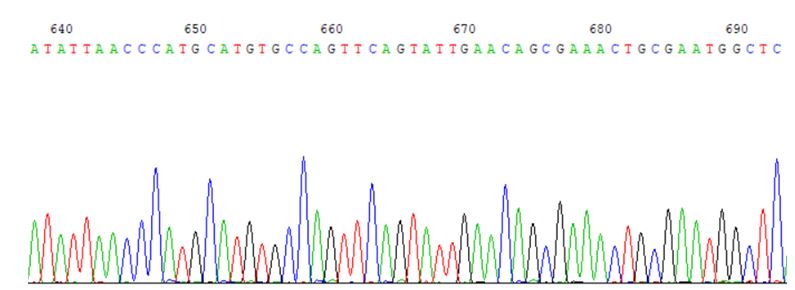
Esempio di elettroferogramma corrispondente ad una sequenza genica.
Risultati
Isolamento dei ceppi
Le condizioni di laboratorio, a volte, possono risultare ostili per alcuni microrganismi. Per questo motivo, in questo studio non è stato possibile mantenere in collezione tutte le colture monoclonali ottenute dagli isolamenti iniziali.
Inizialmente, da 2 dei 3 campioni prelevati nel sito della Grotta dei Laghi (sigla Agn), 1Agn e 2Agn, sono state isolate un elevato numero di cellule (che hanno dato origine ad altrettanti ceppi) in quanto, a una prima analisi allo stereo-microscopio, erano stati osservati due forme diverse di protisti, una di dimensioni maggiori rispetto all’altra. Durante il mantenimento in coltura, nella maggior parte dei ceppi, si è osservata la comparsa di una forma in un isolato dove questa non era presente inizialmente, finendo per avere nella solita provetta entrambe le forme. La comparsa e scomparsa della forma grande si è osservata essere particolarmente evidente a seguito dell’aggiunta o meno di nutrimento.
Per l’allestimento di colture monoclonali da mantenere in collezione sono stati selezionati 12 ceppi. Su 4 di questi ceppi (1Agn3, 1Agn18, 2Agn7 e 2Agn15), selezionati in base al numero e motilità delle cellule e alla presenza della forma grande e piccola, sono state condotte le analisi morfologiche e genetiche: nei ceppi 1Agn18 e 2Agn15 fin dall’isolamento è stata osservata solamente la presenza della forma piccola; nei ceppi 1Agn3 e 2Agn7 è stata osservata la comparsa della forma grande a partire da un isolato della forma piccola.
I ceppi inizialmente isolati dal terzo campione (3Agn) non sono stati in grado di adattarsi alle condizioni di laboratorio.
Dai campioni prelevati nei siti della Buca della cima del Monte Matanna (sigla Mat), dalla Grotta n.1 di Poggio Pinzo (sigla Pin) e dalla Grotta dei Ladri (sigla Lad) non sono state ottenute colture stabili in quanto i ceppi inizialmente isolati hanno richiesto tempi maggiori per adattarsi alle condizioni di laboratorio e sono attualmente in fase di stabilizzazione.
Sui ceppi 1Mat3, 1Mat6, 1Pin4 e 1Pin5 dei primi due siti di campionamento sono state svolte le analisi morfologiche.
Dal sito della Buca del Pierpi (sigla Pie) non sono stati isolati ceppi.
Analisi morfologiche
Le analisi morfologiche, per determinare almeno il genere a cui ciascun ceppo appartiene, sono state condotte attraverso un’indagine eseguita con l’applicazione di immagini fotografiche. Alcune specie di protisti mostrano caratteristiche distintive ed evidenti, mentre altre specie hanno forma e strutture simili a specie appartenenti allo stesso genere o anche a generi filogeneticamente correlati: in questi casi è necessaria un’analisi molecolare aggiuntiva per ottenere risposte univoche sull’identificazione tassonomica a livello di specie.
Grotta dei Laghi (ceppi 1Agn e 2Agn)
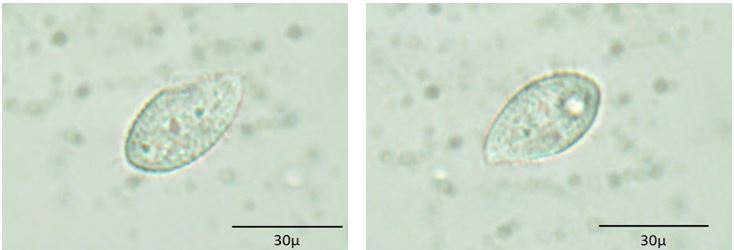
Le cellule isolate (Fig. 7) hanno una forma ovale, più allungata, e la presenza di cilia lungo tutto il contorno cellulare. All’interno delle cellule si osserva la presenza di formazioni vacuolari e, nella forma grande, il citoplasma assume un colore verde scuro. Le dimensioni della forma piccola sono comprese tra i 30 e i 40 μm, mentre la forma grande presenta dimensioni comprese tra i 50 e i 60 μm. Non è stato possibile determinare il genere di appartenenza a causa di caratteri morfologici particolarmente diffusi in vari raggruppamenti tassonomici.
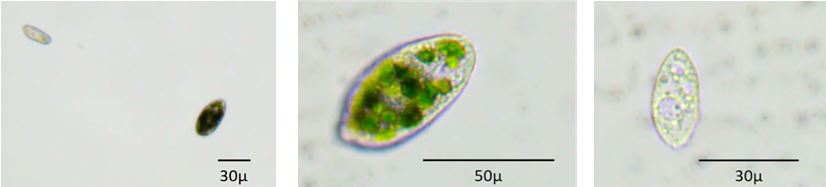
Cellule isolate nella Grotta dei Laghi; ingrandimenti 10x, 40x.
Buca della cima del Monte Matanna (ceppi 1Mat)
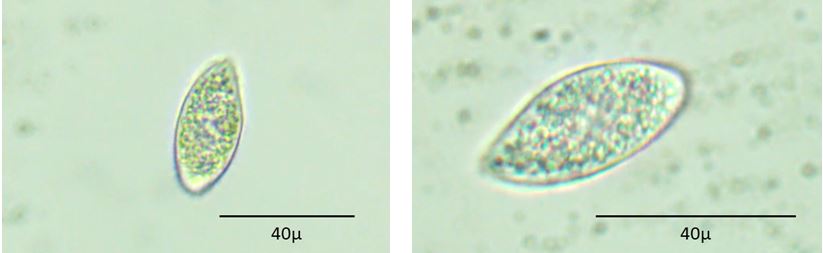
Le cellule isolate (Fig. 8) presentano una forma ovale con cilia lungo tutto il contorno cellulare. All’interno delle cellule, si osservano formazioni vacuolari. La dimensione più lunga delle cellule è di circa 30 μm. Non è stato possibile determinare il genere di appartenenza a causa di caratteri morfologici particolarmente diffusi in vari raggruppamenti tassonomici.
Cellule isolate nella Buca della cima del Monte Matanna; ingrandimenti 10x, 40x.
Grotta n.1 di Poggio Pinzo (ceppi 1Pin)
Le cellule isolate (Fig. 9) presentano una forma ovale con cilia lungo tutto il contorno cellulare. All’interno delle cellule, si osserva un citoplasma di color verde. Le dimensioni delle cellule sono comprese tra i 40 e i 50 µm. Non è stato possibile determinare il genere di appartenenza a causa di caratteri morfologici particolarmente diffusi in vari raggruppamenti tassonomici.
Cellule isolate nella Grotta n.1 di Poggio Pinzo; ingrandimenti 10x, 40x.
Identificazione genetica
L’amplificazione genetica del gene 18S dei campioni mediante la tecnica della PCR ha permesso di ottenere, nei 4 ceppi selezionati per le analisi genetiche (1Agn3, 1Agn18, 2Agn7 e 2Agn15), un prodotto della dimensione attesa di circa 1.800 pb per tutti i ceppi analizzati.
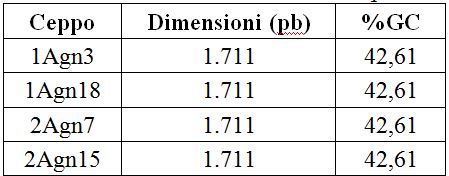
In tabella 3 sono riportati le dimensioni e il contenuto percentuale in GC delle sequenze ottenute: come si può osservare tutte le sequenze presentano una stessa dimensione ed uno stesso contenuto in GC.
Tab. 3: Dimensioni e contenuto in GC delle sequenze ottenute.
Una prima analisi comparativa delle sequenze ottenute mediante allineamento delle stesse ha mostrato che alcune sequenze sono risultate identiche tra loro; pertanto, le 4 sequenze ottenute sono state suddivise in 2 gruppi di identità di sequenza che differiscono tra loro per 2 mutazioni nucleotidiche: un gruppo di sequenza costituito da quelle ottenute dai ceppi 1Agn3, 1Agn7 e 2Agn15 ed un altro gruppo costituito dalla sola sequenza ottenuta dal ceppo 1Agn18. Successivamente, ciascuno dei 2 diversi gruppi di sequenze così ottenute è stato comparato con sequenze omologhe disponibili nel database molecolare, prendendo in considerazione, per ciascuna sequenza nucleotidica ottenuta, quelle che presentavano la maggiore percentuale di identità di sequenza con le specie da noi isolate. I valori di queste analisi sono riportati nella tabella 4.
Tab. 4: Risultati delle analisi comparative delle sequenze ottenute con quelle disponibili nel database molecolare.
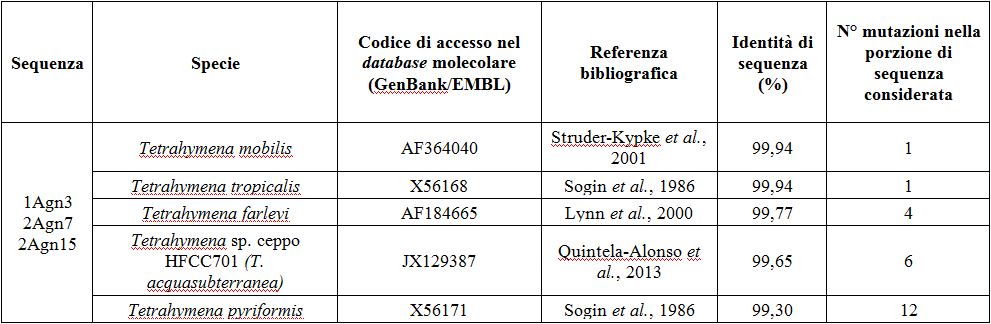
Il ceppo 1Agn18 differisce per lo stesso numero di mutazioni nucleotidiche del primo gruppo di identità di sequenza con le specie citate in tabella 4, eccezion fatta per Tetrahymena farleyi da cui differisce per 5 mutazioni. L’analisi comparativa delle sequenze ottenute dai ceppi di entrambi i gruppi di sequenze con sequenze omologhe presenti nel database molecolare ha mostrato il più alto valore di identità con sequenze appartenenti a specie del genere Tetrahymena, in particolare T. mobilis e T. tropicalis. Entrambe queste specie rientrano all’interno del clado “borealis”, uno dei gruppi in cui sono suddivise le specie appartenenti al genere Tetrahymena (Struder-Kypke et al., 2001).
Conclusioni
Le analisi di questo studio si basano sull’identificazione e caratterizzazione morfologiche e genetiche di protisti ciliati isolati da aree ipogee della regione Toscana.
Per tale scopo sono state prese in considerazione 5 aree di studio: Buca della cima del Monte Matanna, nel comune di Stazzema (Lucca); Grotta dei Laghi e Grotta dei Ladri, nel comune di Agnano (Pisa); Buca del Pierpi, nel comune di Murlo (Siena); Grotta n.1 di Poggio Pinzo, nel comune di Monte Argentario (Grosseto).
Nel caso dei campioni provenienti dai siti Buca della cima del Monte Matanna, Grotta dei Ladri e Grotta n.1 di Poggio Pinzo, non sono state ottenute colture monoclonali stabili nel tempo in quanto i ceppi isolati hanno richiesto tempi maggiori per adattarsi alle condizioni di laboratorio.
Nel caso del campione proveniente dal sito Buca del Pierpi, non sono stati isolati protisti.
Nel caso dei campioni provenienti dal sito Grotta dei Laghi, sono state ottenute colture monoclonali stabili nel tempo da solo 2 dei 3 campioni raccolti, per un totale di 12 ceppi. Su 4 di questi sono state svolte le analisi genetiche utilizzando un marcatore genetico specie-specifico (gene nucleare per l’RNA ribosomiale 18S), che ha consentito l’identificazione di 2 specie diverse, entrambe appartenenti al genere Tetrahymena. Tali specie, inoltre, differiscono tra loro, nella sequenza del marcatore genetico selezionato (18S), per 2 mutazioni nucleotidiche.
L’analisi comparativa di entrambe le sequenze ottenute dalle 2 specie con quelle omologhe disponibili nel database molecolare ha mostrato un’identità di sequenza pari al 99,9% (con una sola mutazione nucleotidica nella porzione genetica considerata) con specie appartenenti al genere Tetrahymena, in particolare T. mobilis e T. tropicalis. Entrambe queste specie rientrano all’interno del clado “borealis”, uno dei gruppi in cui sono suddivise le specie appartenenti al genere Tetrahymena (Struder-Kypke et al., 2001). Da ricerche bibliografiche, sono state individuate altre specie, appartenenti al sister group di T. mobilis e T. tropicalis, che presentano caratteristiche comuni alle specie da noi isolate. In particolar modo, T. acquasubterranea, isolata da acque sotterranee di Città del Capo (Sudafrica) e caratterizzata da un ciclo vitale bifasico (con la presenza di due forme diverse: una più grande e una più piccola) come quello da noi osservato; e T. farleyi, isolata per la prima volta da un campione di urina di cane e successivamente ritrovata a vita libera in acque sotterranee presso la Stazione di Ricerca Zoologica dell’Università di Cologne (Germania) (Lynn et al., 2000; Quintela-Alonso et al., 2013). Sulla base di questi risultati, è stato ipotizzato che i microrganismi appartenenti alle specie isolate fossero dei parassiti facoltativi, in quanto il sito di campionamento è situato in un’area dove sono presenti animali selvatici e, nelle vicinanze, è presente la Grotta dei Ladri dove al momento vive una colonia di pipistrelli.
In bibliografia, è stato riscontrato uno studio simile finalizzato alla caratterizzazione della comunità protistica delle Grotte di Frasassi (Marche), in cui è stata trovata la specie criptica T. pyriformis, appartenete al clado “borealis”, ma non rientrante nel sister group delle specie precedentemente citate, con cui però condivide un “pyriformis-like life style” (Bharti et al., 2022).
I ceppi provenienti dal sito Grotta dei Laghi sono stati assegnati tassonomicamente al genere Tetrahymena in quanto le sequenze ottenute differiscono per una sola differenza nucleotidica con le corrispondenti sequenze omologhe di riferimento presenti nel database molecolare. Inoltre, si ritiene che le specie isolate siano nuove specie appartenenti al genere Tetrahymena: questo genere presenta un valore di divergenza intraspecifica minore del 1% (Kher et al., 2011); ad esempio T. mobilis e T. tropicalis, due specie strettamente correlate da un punto di vista filogenetico, differiscono tra loro per una sola mutazione nucleotidica.
Il ritrovamento di 2 nuove specie diverse appartenenti al genere Tetrahymena permette di considerare il sito in questione come un promettente laboratorio naturale per la ricerca di specie di protisti produttrici di prodotti naturali, in quanto il genere di protisti identificato è considerato un modello di ricerca per vari tipi di studi, tra cui ricerche sui metaboliti secondari, utilizzabili in diversi settori biotecnologici.
A cura di:
S. Cei, Dipartimento di Biologia, Università di Pisa; F.P. Frontini, Dipartimento di Biologia, Università di Pisa; L. Canale e A. Noferi, Professional Scuba Association International PSAI Italia; G. Di Giuseppe, Dipartimento di Biologia, Università di Pisa; Federazione Speleologica Toscana
Bibliografia e Sitografia
Bharti D., Kumar S., Buonanno F., Ortenzi C., Montanari A., Quintela-Alonso P. & La Terza A. (2022). Free living ciliated protists from the chemoautotrophic cave ecosystem of Frasassi (Italy). Subterranean Biology, 44: 167-198.
Foissner W. (2006). Biogeography and dispersal of microorganisms: a review emphasizing protists. Acta protozoologica, 45(2): 111-136.
Foissner W. & Hawksworth D. (2009). Protist Diversity and Geographical Distribution. Topics in Biodiversity and Conservation. Springer Netherlands.
Hall T.A. (1999). BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT. Nucl. Acids Symp., 41: 95-98.
Hausmann K., Hülsmann N. & Radek R. (2003). Protistology. Schweizerbart’Sche Verlagsbuchhandlung.
Kher C.P, Doerder F.P., Cooper J., Ikonomi P., Achilles-Day U., Küpper F.C. & Lynn D.H. (2011). Barcoding Tetrahymena: discriminating species and identifying unknowns using the cytochrome c oxidase subunit I (cox-1) barcode. Protist, 162(1): 2-13.
Lynn D.H., Gransden S.G., Wright A.-D.G. & Josephson G. (2000). Characterization of a new species of the ciliate Tetrahymena isolated from the urine of a dog: First report of Tetrahymena from a mammal. Acta Protozool., 39: 289-294.
Medlin L., Elwood H., Stickel S. & Sogin M. (1988). The characterization of enzymatically amplified eukaryotic 16S-like rRNA-coding regions. Gene, 71(2): 491-499.
Pawlowski J., Audic S., Adl S., Bass D., Belbahri L., et al. (2012). CBOL Protist Working Group: Barcoding Eukaryotic Richness beyond the Animal, Plant, and Fungal Kingdoms. PLOS Biology. PLoS Biol 10(11): e1001419.
Petroni G., Dini F., Verni F. & Rosati G. (2002). A molecular approach to the tangled intrageneric relationships underlying phylogeny in Euplotes (Ciliophora, Spirotrichea). Mol. Phylogenet. Evo., 22(1): 118-130.
Quintela-Alonso P., Nitsche F., Wylezich C., Arndt H. & Foissner W (2013). A new Tetrahymena (ciliophora, oligohymenophorea) from groundwater of cape town, South Africa. J. Eukaryot. Microbiol., 60 (3): 235-246.
Sogin M.L., Ingold A., Karlok M., Nielsen H. & Engberg J. (1986). Phylogenetic evidence for the acquisition of ribosomal RNA introns subsequent to the divergence of some of the major Tetrahymena groups. EMBO J., 5(13): 3625-3630.
Struder-Kypke M.C., Wright A.D., Jerome C.A. & Lynn D.H. (2001). Parallel evolution of histophagy in ciliates of the genus Tetrahymena. BMC Evol. Biol., 1(1): 5.
Thompson J.D., Gibson T.J., Plewniak F., Jeanmougin F. & Higgins D.G. (1997). The ClustalX windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucl. Acids Res., 4: 4876-4882.
Database molecolare GenBank: www.ncbi.nlm.nih.gov
Associazione Professional Scuba Association International (PSAI) Italia: www.psai.it
Gruppo Speleologico Torrentistico Speolo (GSTS): www.speolonet.wordpress.com
Federazione Speleologica toscana https://www.speleotoscana.it/